单细胞数据去批次效应算法比较
来源:**BatchBench**和生信技能树jimmy
批次整合效果
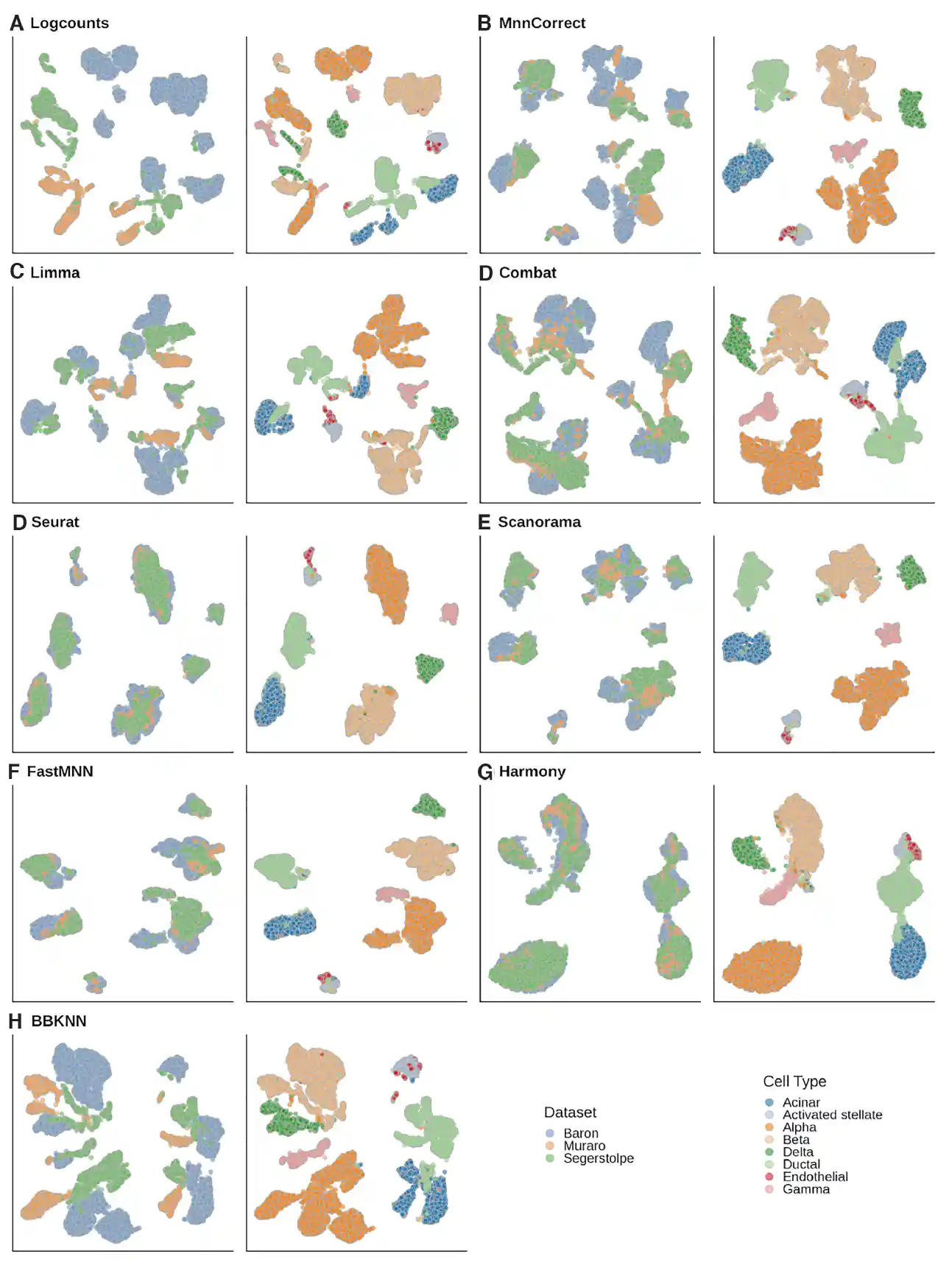
- 左图是批次上色,右图是细胞类型上色
- 左图不同区域颜色越混杂越好
- 右图不同区域颜色越纯粹越好,且同一颜色最好在且只在一个区域
资源消耗量
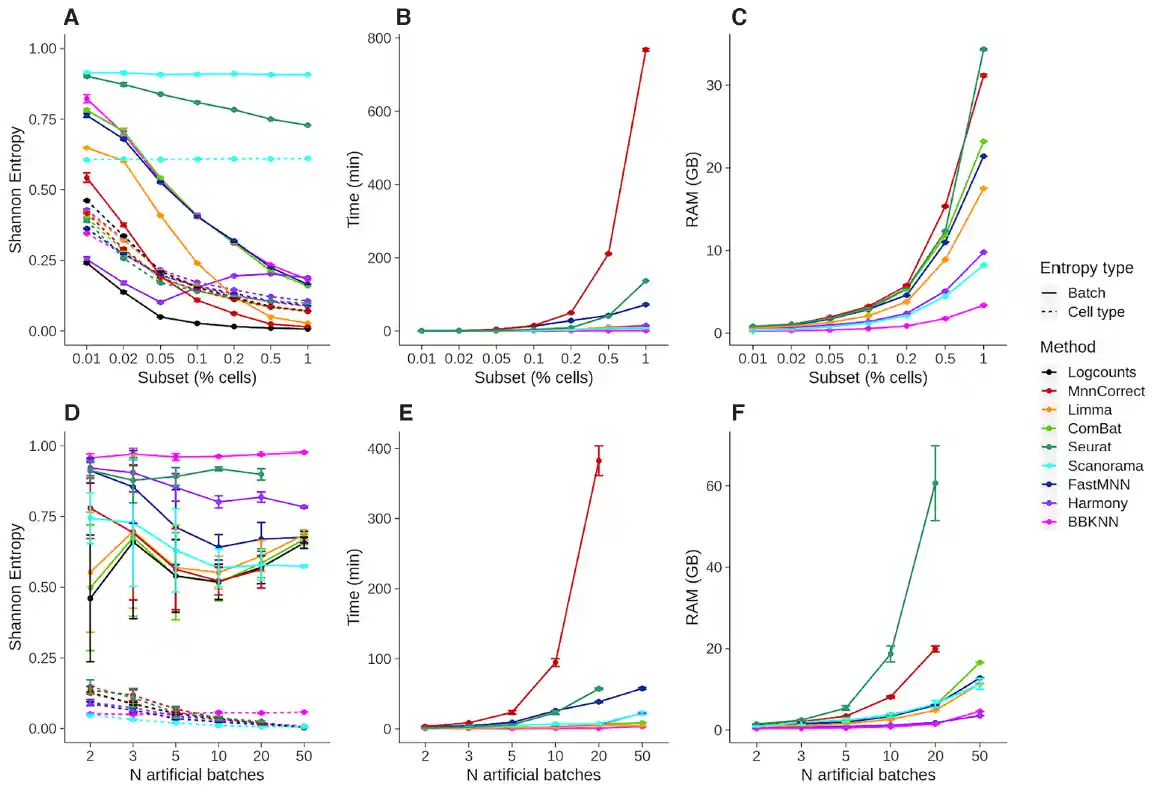
- 实线是批次的熵,越高表示混合程度越好
- 虚线是细胞类型的熵,越低表示区分度越好
- 第一行是批次数量固定为2,细胞数量增长(总计61,000进行下采样)
- 第二行是每个批次固定1001个细胞,批次数量增长
- 第二列是计算消耗的时间,不清楚作者的计算节点性能,但应该很强
- 第三列是计算占用的内存,可以看到Seurat在2个批次6万个细胞时占用了高于30G的内存;而20个批次,2万个细胞时占用了60G内存
校正批次后对聚类的影响
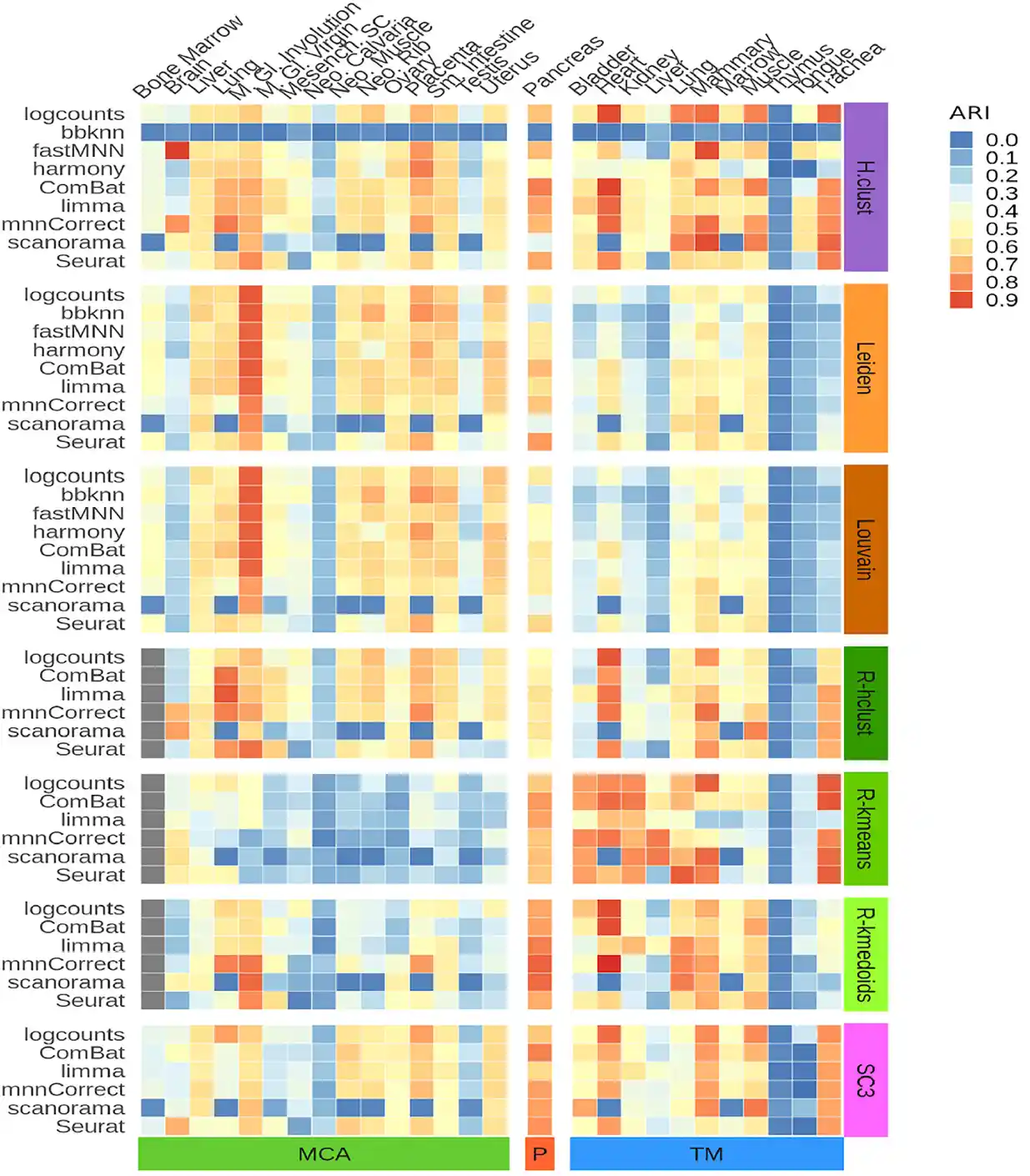
- 上面的col是组织,下面的col是数据来源,左边的row是批次去除算法,右边的row是无监督聚类算法
- 颜色表示调整兰德指数的值,越接近1越好,说明该组织越适合这种批次去除算法和无监督聚类算法
校正批次后对Marker基因的影响
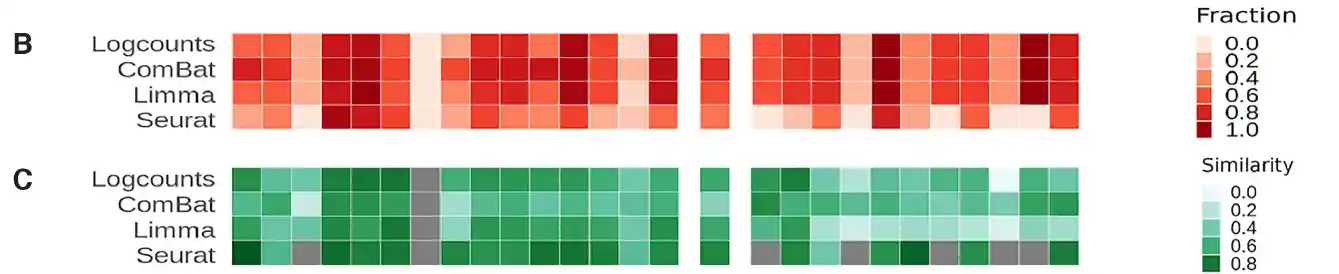
- 红色表示DEG中检测到的标记基因数量占实际标记基因数量的比例
- 深青色表示DEG与实际标记基因集合的相似性
- 可以看出效果一塌糊涂,还是先分群再合并比较好,这些校正批次效应的算法只能用于可视化。
单细胞数据去批次效应算法比较
https://b.limour.top/1970.html