TCGA (一) 获取数据
第一步 安装程辑包并加载
1 | |
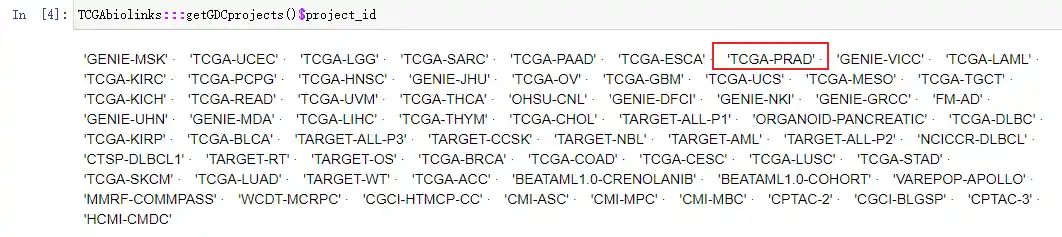
第二步 查看癌症类型
1 | |
癌症类型说明:癌症类型和样本代号详解TCGA
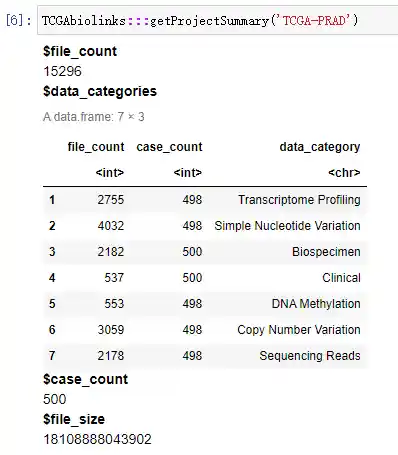
第三步 查看对应癌症的数据类型
1 | |
case_count为病人数,file_count为对应的文件数,”Transcriptome Profiling”表示表达谱
“如何玩转生物大数据”系列:TCGA的样本注释信息和数据类型统计
1)转录组数据(Transcriptome)
2)甲基化数据(Methylation)
3)基因突变数据(Mutation)
4)拷贝数变化数据 (CNV)
TCGAbiolinks包下载TCGA数据进行表达差异分析-乳腺癌案例
(4)workflow.type
该数据类型有很多种,根据data.type的不同而不同,不同的数据类型,有其对应的参数可供选择。比如Gene Expression Quantification数据类型下workflow.type 有4种类型分别为:
HTSeq - FPKM-UQ:FPKM上四分位数标准化值
HTSeq - FPKM:FPKM值/表达量值
HTSeq - Counts:原始count数
STAR - Counts
具体可在GDC官网查看
(5)legacy
这个参数主要是因为TCGA数据有两个入口可以下载,GDC Legacy Archive 和 GDC Data Portal,区别主要是注释参考基因组版本不同分别是:GDC Legacy Archive(hg19和GDC Data Portal(hg38)。参数默认为FALSE,下载GDC Data Portal(hg38)。这里建议是,下载转录组层面的数据使用hg38,下载DNA层面的数据使用hg19,因为比如做SNP分析的时候很多数据库没有hg38版本的数据,都是hg19的。
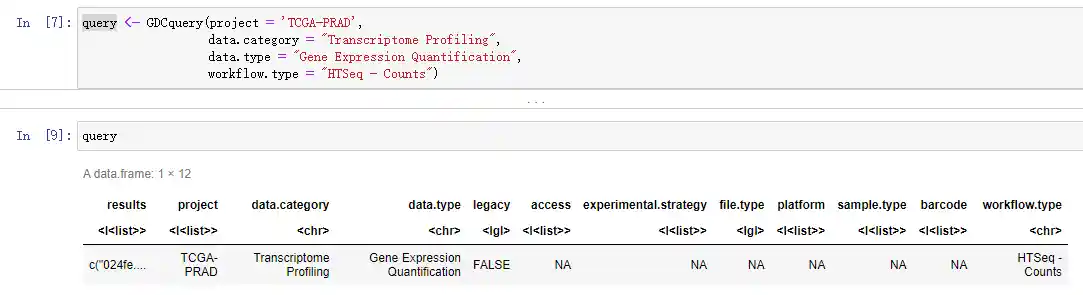
第四步 下载对应数据
1 | |
第五步 保存对应数据
1 | |
TCGA (一) 获取数据
https://b.limour.top/770.html